实例4:蛋白质信息分析
蛋白质(protein)是生命的物质基础,是组成生物体一切细胞、组织的重要成分,机体所有重要的组成部分和活动都需要有蛋白质的参与。
 |
 |
 |
蛋白质由氨基酸组成,不同蛋白质的长度、氨基酸组成、等电点不同,并且形成不同的二级结构、三级结构(空间结构)甚至四级结构(多个亚基的蛋白)。
生物信息学分析工具包EMBOSS提供了多种分析蛋白质性质的工具。
首先我们先看一个比较简单的序列信息分析程序infoseq,该程序统计核酸和蛋白质序列的名称、登录号、长度等信息。我们用该程序统计上一节用的蛋白质序列的长度:
[xiezy@ibi98 ml]$ infoseq -only -name -length lde.fa
Display basic information about sequences
Name Length
Musmu 550
Homsa 544
Anoga 558
Caeel 500
Drome 534
Danre 568
Xentr 534
pepstats是分析统计蛋白质的分子量、等电点、电荷及氨基酸组成信息的程序:
[xiezy@ibi98 ml]$ pepstats lde_hs.fa Calculate statistics of protein properties Pepstats program output file [homsa.pepstats]: [xiezy@ibi98 ml]$ cat homsa.pepstats PEPSTATS of Homsa from 1 to 544 Molecular weight = 61554.93 Residues = 544 Average Residue Weight = 113.152 Charge = -16.0 Isoelectric Point = 5.1441 A280 Molar Extinction Coefficients = 67270 (reduced) 67895 (cystine bridges) A280 Extinction Coefficients 1mg/ml = 1.093 (reduced) 1.103 (cystine bridges) Probability of expression in inclusion bodies = 0.639 Residue Number Mole% DayhoffStat A = Ala 36 6.618 0.769 B = Asx 0 0.000 0.000 C = Cys 11 2.022 0.697 D = Asp 42 7.721 1.404 E = Glu 44 8.088 1.348 F = Phe 16 2.941 0.817 G = Gly 33 6.066 0.722 H = His 22 4.044 2.022 I = Ile 30 5.515 1.225 J = --- 0 0.000 0.000 K = Lys 32 5.882 0.891 L = Leu 47 8.640 1.168 M = Met 10 1.838 1.081 N = Asn 23 4.228 0.983 O = --- 0 0.000 0.000 P = Pro 28 5.147 0.990 Q = Gln 22 4.044 1.037 R = Arg 27 4.963 1.013 S = Ser 39 7.169 1.024 T = Thr 24 4.412 0.723 U = --- 0 0.000 0.000 V = Val 29 5.331 0.808 W = Trp 6 1.103 0.848 X = Xaa 0 0.000 0.000 Y = Tyr 23 4.228 1.244 Z = Glx 0 0.000 0.000 Property Residues Number Mole% Tiny (A+C+G+S+T) 143 26.287 Small (A+B+C+D+G+N+P+S+T+V) 265 48.713 Aliphatic (A+I+L+V) 142 26.103 Aromatic (F+H+W+Y) 67 12.316 Non-polar (A+C+F+G+I+L+M+P+V+W+Y) 269 49.449 Polar (D+E+H+K+N+Q+R+S+T+Z) 275 50.551 Charged (B+D+E+H+K+R+Z) 167 30.699 Basic (H+K+R) 81 14.890 Acidic (B+D+E+Z) 86 15.809
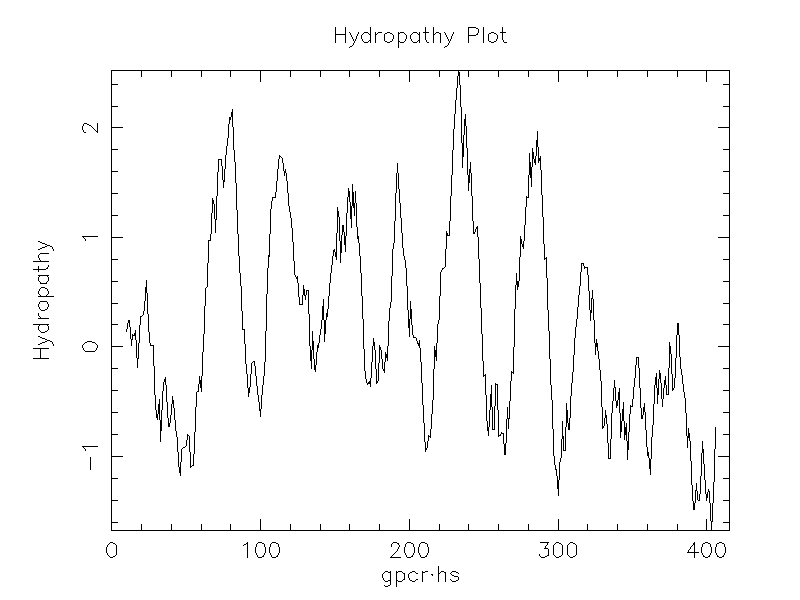
pepwindow程序用来分析蛋白质亲水性并画图,除了直接显示结果外,还可以输出多种格式的图像文件。如用该程序分析人的G蛋白偶联受体(G-protein coupled receptor, GPCR)的亲水性并输出成PNG格式的图像文件:
[xiezy@ibi98 ml]$ pepwindow GPCR_hs.fa
Draw a hydropathy plot for a protein sequence
Graph type [x11]: png
Created pepwindow.1.png
将图像文件pepwindow.1.png下载到本地查看,可以发现GPCR标志性的7次跨膜结构。
EMBOSS中还有很多蛋白质分析相关的程序,这些命令行工具用起来不如拥有图形界面的软件直观,但可以实现自动化和批量化运行,应用熟练后可以极大提高数据处理和分析的效率。
到这里,整个课程就接近尾声了。通过前面的学习,我们可以发现,shell就像一个大飞机的组装厂。大飞机的生产厂家尽管自己不能生产发动机等配件(或者自己生产的不如专业的厂家生产的好),但可以通过组合不同厂家生产的优质的配件,生产出高质量的大飞机。shell本身的效率不高,但可以方便地使用shell命令或其他语言写好的程序,把他们通过管道或控制结构组合起来,用少量的代码就可以完成复杂的任务。